드물게 있는 검사 중 하나인 EBV 정량 검사. 족보대로 판독을 하는거는 쉬운데, Real time-PCR을 이해를 할려고 하니 어렵다. ㅡㅡ
좀 이상한 부분이 있다면 PCR 소트트웨어가 읽은 값을 바탕으로 내가 직접 선형회귀분석을 한 후 원하는 값을 구하는 과정을 진행해보면 내가 구한 값과 소프트웨어가 구한 값이 약간 차이가 있다는 것이다. 뭐가 문제일까…
# 자료 입력
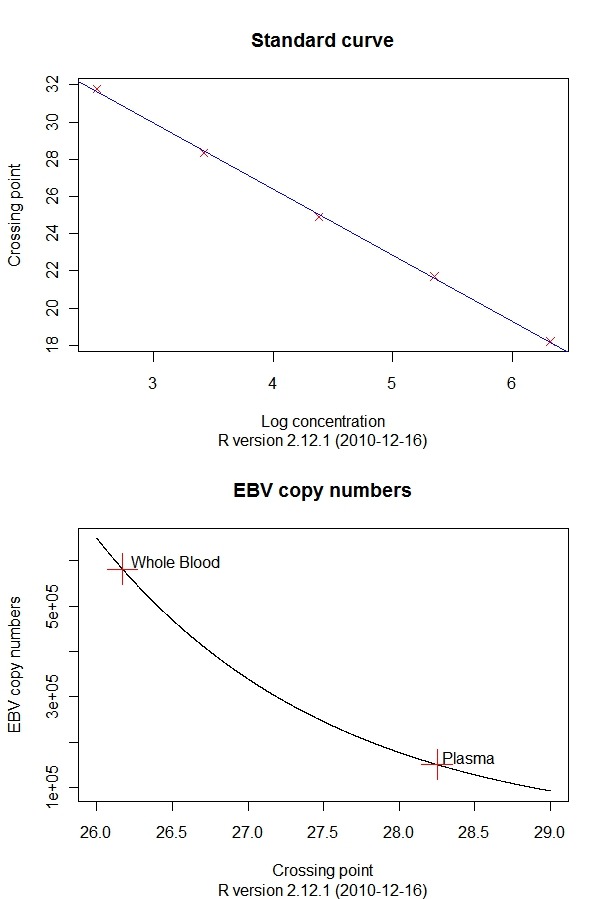
cp <- c(18.21,21.70,24.92,28.32,31.79)
concentration <- c(2.08e6,2.23e5,2.41e4,2.63e3,3.35e2)
#밑이 10인 로그값을 사용한다.
logconc <- log10(concentration)
#선형회귀함수를 구한다.
standard <- lm(cp ~ logconc)
#이 방법으로 그래프 화면을 2개로 나누어서 볼 수 있다.
par(mfrow=c(2,1))
#Standard curve 를 그리고
plot(cp~logconc, xlab=”Log concentration”,ylab=”Crossing
point”,main=”Standard curve”,type=”p”, col=”red”, lwd=1, pch=4,
sub=R.version.string)
#프로그램이 구해준 선형함수를 그린다.
abline(standard, col=”blue”)
#y=a+bx 에서 a, b 값은 다음과 같이 추출할 수 있다.
a <- standard$coefficients[1]
b <- standard$coefficients[2]
#아직 좋은 방법을 찾지는 못했고, EBV 정량검사를 위한 수식을 구한 것. 현재 보고 방식으로는 50배 곱한 수치가 필요함.
x=seq(26,29, by=0.001) ; 그래프를 그리기 위한 x 값의 범위 지정
y=10^((x-a)/b)*50 ; y 값을 구하기 위한 수식 지정
#이제 검사 검체의 CP 값에 따른 추정 EBV 정량검사를 알 수 있다.
plot(x,y, type=”l”, main=”EBV copy numbers”, xlab=”Crossing point”, ylab=”EBV copy numbers”, sub=R.version.string)
#우리가 구하고자 하는 값의 CP 값.
x <- c(26.17, 28.25); y <- 10^((x-a)/b)*50
points(x[1],y[1], col=”red”, cex=3, pch=3);text(x[1],y[1],”Whole Blood”, adj=c(-.1,-.1))
points(x[2],y[2], col=”red”, cex=3, pch=3);text(x[2],y[2],”Plasma”, adj=c(-.1,-.1))