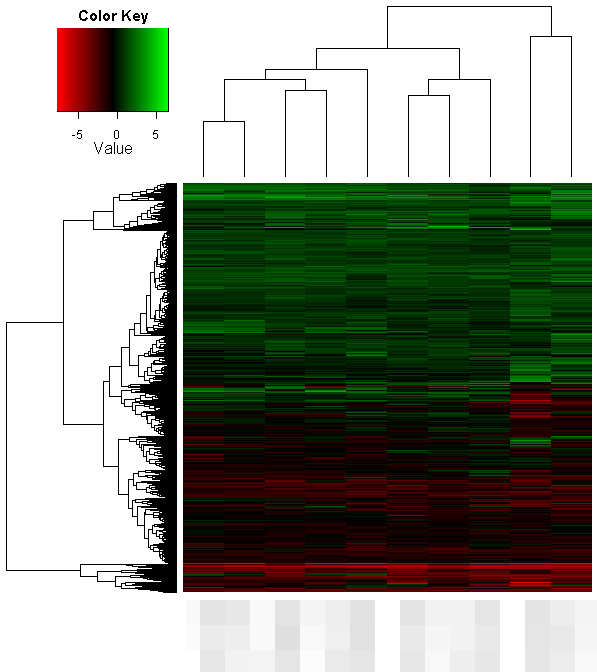
지난번 Heatmap 결과(2010/05/01 – [공부해 봅시다/R-Project] – Heatmap 만들기) 를 통하여 앞의 2개의 증례 가 다른 증례와는 다소 차이가 나는 것을 확인해 보았다. 하지만 Heatmap 을 만드는 과정에서 자료의 위치가 이동하기 때문에 원자료를 찾아보면 그 2개의 증례는 10번, 11번째 column 에 위치하는 것을 확인하였다.
여기서 부터가 삽질이었다. 특정 질환에서 발현이 증가하고 다른 질환에서 발현이 감소하는 그러한 값을 찾는것이 기본이 되기 때문에 이 미묘한 자료를 찾는 것은 엑셀에서 순차 정렬을 11번 반복하게 된다. OTL
게다가 cut-off 값을 바꾸면 바꿀 때 마다 이 짓을 계속 해야하기 때문에 인터넷 바다를 검색한 끝에 해결책을 찾았다. 비록 결과물이 좀 보기 흉하기는 하지만, 내가 할 삽질을 컴퓨터가 대신 하도록 하는 방법을 찾을 수는 있었다.
data <- read.table(“b08.csv”, header=T, sep=”,”)
설명) 자료를 불러오는 것. 이 자료는 지난 자료와는 달리 첫 column 에 chip의 설명을 담고 있다. 특정 유전자를 찾기 위해서는 필요한 정보이다. 문자값이 있기 때문에 Heatmap 으로 진행은 할 수가 없다.
x <- which(data[,2] < X & data[,3] < X & data[,4] < X & data[,5] < X & data[,6] < X & data[,7] < X & data[,8] < X & data[,9] < X & data[,10] < X & data[,11] >= X & data[,12] >= X)
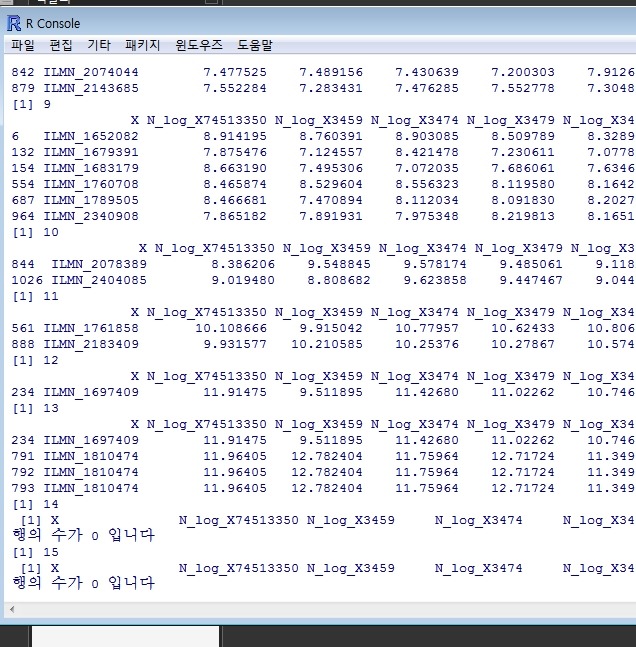
설명) 식이 좀 지저분하지만 Ctrl+C 와 Ctrl+V를 반복하면 쉽게 만들 수 있다. data 의 2~10 column 까지는 X 값보다 작은 것을 선택하고, 11, 12 column 은 X 값보다 같거나 큰 값을 선택한다. 아직 정확한 사용법을 알고 있지는 않지만 이렇게 한 후에 x 값을 살펴 보면 row 번호만 나왔다. 그래서 다음과 같은 식을 입력해주면 그 row 에 해당하는 값을 볼 수가 있다.
data[x,]
X 값을 1부터 대략 20 정도까지 일일이 입력하고 그 결과를 확인하면 되지만.. 그래도 반복되는 구문이라서 해결책을 찾을 수 있었다.
for (X in 1:20) {
x <- which(data[,2] < X & data[,3] < X & data[,4] < X & data[,5] < X & data[,6] < X & data[,7] < X & data[,8] < X & data[,9] < X & data[,10] < X & data[,11] >= X & data[,12] >= X)
print(X);print(data[x,])}
설명) 역시 R-project의 문법을 정확하게 이해하고 있지 않아서 생긴 지저분한 문장이다. {} 괄호로는 하나의 식을 입력할 수 있고, 엔터키로 문장을 바꾸면 실행을 하게 되는 것 같기는 한데.. ㅡㅡ;;
X가 1에서 20까지 1씩 증가하는 값으로 되어 있고 그에 따라서 x 값이 나온다. 결과를 보여주기 위해서 어떤 X 값인지 보여주는 print(X) 와 그 X 값에 따른 결과물을 볼 수가 있다.