 금요일에 세포병리학회 참석으로 인하여 연세대학교병원에 갔다. 외래쪽 건물의 6층에 있어도 3층에서 하는 노래 소리가 잘 들린다는 점은 참 좋았다.
금요일에 세포병리학회 참석으로 인하여 연세대학교병원에 갔다. 외래쪽 건물의 6층에 있어도 3층에서 하는 노래 소리가 잘 들린다는 점은 참 좋았다.
 연세대학교병원에 물이 졸졸 흐르는 곳이 있었는데 물이 조금 세차게 흐른다는 느낌이 들었다. 조금 느리게 흘렀다면 좋았을 것 같기도 하고..
연세대학교병원에 물이 졸졸 흐르는 곳이 있었는데 물이 조금 세차게 흐른다는 느낌이 들었다. 조금 느리게 흘렀다면 좋았을 것 같기도 하고..

 금요일에 세포병리학회 참석으로 인하여 연세대학교병원에 갔다. 외래쪽 건물의 6층에 있어도 3층에서 하는 노래 소리가 잘 들린다는 점은 참 좋았다.
금요일에 세포병리학회 참석으로 인하여 연세대학교병원에 갔다. 외래쪽 건물의 6층에 있어도 3층에서 하는 노래 소리가 잘 들린다는 점은 참 좋았다.
 연세대학교병원에 물이 졸졸 흐르는 곳이 있었는데 물이 조금 세차게 흐른다는 느낌이 들었다. 조금 느리게 흘렀다면 좋았을 것 같기도 하고..
연세대학교병원에 물이 졸졸 흐르는 곳이 있었는데 물이 조금 세차게 흐른다는 느낌이 들었다. 조금 느리게 흘렀다면 좋았을 것 같기도 하고..
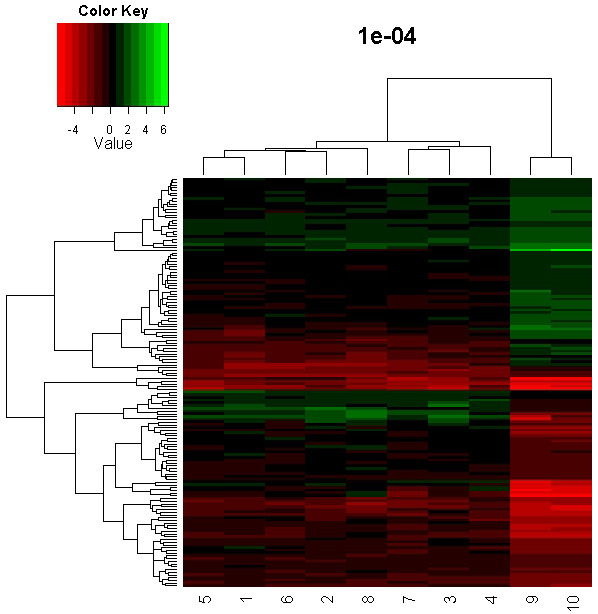

오늘 아침에 Journal 발표 시간에 갑자기 생각난 아이디어를 바탕으로 heatmap 을 다시 만들어 보았다. 만들면서 느낀 건데 이런 chip 기반의 data는 해석하는데 항상 주의가 필요하다. 원 데이터를 조작하지 않아도 중간의 과정을 저자가 원하는 방향으로 이끌게 되면 그렇게 보이는 자료를 만들 수 있다. 🙂 이 heatmap 은 9, 10 번과 그 이외의 자료가 유의한 차이를 나타나도록 만든 것이다. 하지만, 마음만 먹으면 다른 자료가 유의하게 나오도록 한 다음 다른 설명을 하는 것도 가능하다. -_-
그림 만드는 것은 어느정도 해결이 되었으니 이제는 원자료를 해석하는데에 집중해야겠다.
그렇게 깔끔한 구문이라고는 못하지만 문법적인 문제는 없는 것 같고, 컴퓨터도 그렇게 느린 편은 아니라서 좀 길게 만들어도 신경을 쓰지 않았다.
2010/05/24 – [공부해 봅시다/R-Project] – Green-Black-Red heatmap
2010/05/26 – [공부해 봅시다/R-Project] – Array data 살펴 보기 – Simple
library(gplots)
data <- read.table(“Book1-1-3.csv”, header=T, sep=”,”)
CONF <- 0.0001 #유의수준
COUNT <- 0 #유의한 값을 가지는 항목 숫자세기
for (X in 1:5467) {
x <- c(data[X,2],data[X,3],data[X,4],data[X,5], data[X,6],data[X,7],data[X,8],data[X,9])
y <- c(data[X,10],data[X,11])
z1 <- t.test(x,y, var.equal=FALSE, paired=FALSE, conf.level=CONF)
z2 <- t.test(x,y, var.equal=TRUE, paired=FALSE, conf.level=CONF)
if (z1$p.value < CONF | z2$p.value < CONF) {COUNT <- COUNT +1}
}
newdata <- matrix(nrow=COUNT, ncol=10) #유의한 값을 가지는 것을 위한 새로운 matrix 만들기
COUNT1 <- 0
for (X in 1:5467) {
x <- c(data[X,2],data[X,3],data[X,4],data[X,5], data[X,6],data[X,7],data[X,8],data[X,9])
y <- c(data[X,10],data[X,11])
z1 <- t.test(x,y, var.equal=FALSE, paired=FALSE, conf.level=CONF)
z2 <- t.test(x,y, var.equal=TRUE, paired=FALSE, conf.level=CONF)
if (z1$p.value < CONF | z2$p.value < CONF) {COUNT1 <- COUNT1 +1
newdata[COUNT1, 1] <- data[X,2];newdata[COUNT1, 2] <- data[X,3];newdata[COUNT1, 3] <- data[X,4];newdata[COUNT1, 4] <- data[X,5];
newdata[COUNT1, 5] <- data[X,6];newdata[COUNT1, 6] <- data[X,7];newdata[COUNT1, 7] <- data[X,8];newdata[COUNT1, 8] <- data[X,9];
newdata[COUNT1, 9] <- data[X,10];newdata[COUNT1, 10] <-data[X,11]
}
}
my.hclust <- function(x){hclust(x, method=”complete”)}
my.dist <- function(x){dist(x, method=”euclidean”)}
heatmap.2(dat, col=rainbow(20*10, start = 0/6, end = 4/6), hclustfun=my.hclust, distfun=my.dist, dendrogram=”both”, trace=”none”, density.info = “none”, key = TRUE)


KTX Magazine 인가 하는 곳에서 이 책을 소개하고 있어서 한 번 사서 봤다. 저자는 엄청나게 꼼꼼히 Gross를 하는지 정말 구석수석에 있는 포인트를 잡아내는 능력이 있다. 그리고 불교 지식에 해박하여 설명도 자세하다. 불교에 대한 지식이 전혀 없다면 오히려 이 책을 한 번쯤 읽어보는 것이 좋을 것 같다.