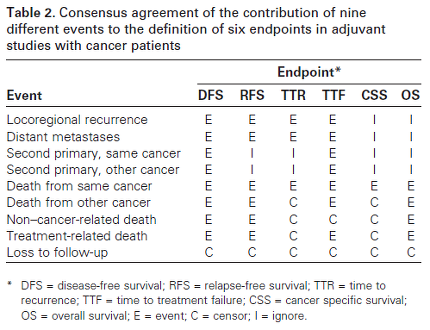
여러 논문을 찾아본 결과, 2007년 Punt 의 논문이 대장암 연구에서 사용되는 각종 survival time 에 대한 정의를 내려놓은 것을 찾을 수 있었다. 이 방식을 다른 암종에도 적용할 수 있는 것은 아니겠지만, 참고할 수는 있으니 꼭 알아두어야 할 논문이라고 할 수 있을 듯 하다. 이 외에 유방암에 대하여 정의해둔 논문이 있기는 한데(J Clin Oncol. 2007;25(15):2127-32.), 이 논문은 내가 쓸 일은 없을 듯 하니 이 번에는 생략함.
DFS의 경우 좀 유의할 만한 점은 다른 암이 생겼거나 다른 이유로 죽어도 모두 event 로 정의한 다는 것이다. 상당히 의외의 부분이었음. 생존 분석을 할 때에 굉장히 구체적으로 자료를 미리 정해두는 것의 필요성을 알게 해주었음.
Reference: Punt CJ, Buyse M, Kohne CH, Hohenberger P, Labianca R, Schmoll HJ, et al. Endpoints in adjuvant treatment trials: a systematic review of the literature in colon cancer and proposed definitions for future trials. J Natl Cancer Inst. 2007;99(13):998-1003.